ゲノム情報がなかったアセロラも10本の染色体に対応する塩基配列を解読

画像はイメージです
かずさDNA研究所とニチレイフーズ、鹿児島大学は共同でアセロラの全ゲノムを解読したことを発表した。
イラストはイメージです
アセロラは中南米やカリブ諸島が原産の熱帯性低木で、ブラジルやベトナムで生産されている。果実に天然ビタミンCが豊富に含まれることから、果汁やパウダー等に加工され食品原料や化粧品原料、クリーンラベルとして注目を集めている。
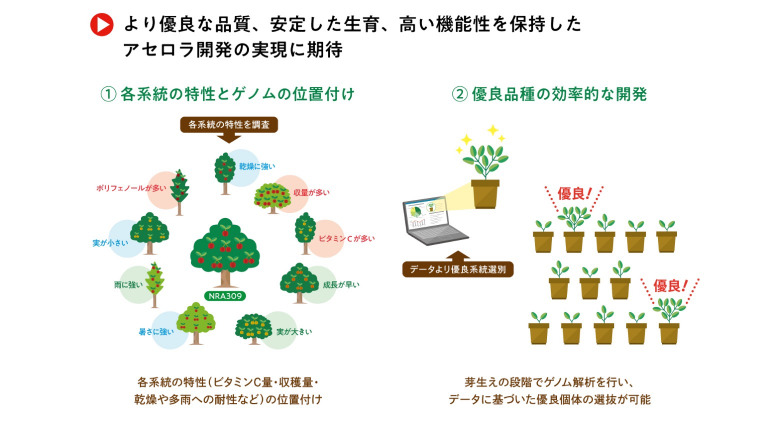
近年は、より高い天然ビタミンCやより特性の良いアセロラ品種の開発が求められているが、アセロラでは効率の良い品種開発に十分なゲノム情報が得られていなかった。
■ゲノム情報を活用して品種改良に利用されているアセロラの遺伝的な違いを解明
そこで本研究では、最新のロングリード技術を利用してアセロラのゲノム解読を行なうと同時に、取得したゲノム情報を活用して品種改良に利用されているアセロラの系統の遺伝的な違いを明らかにすることを目的とした。
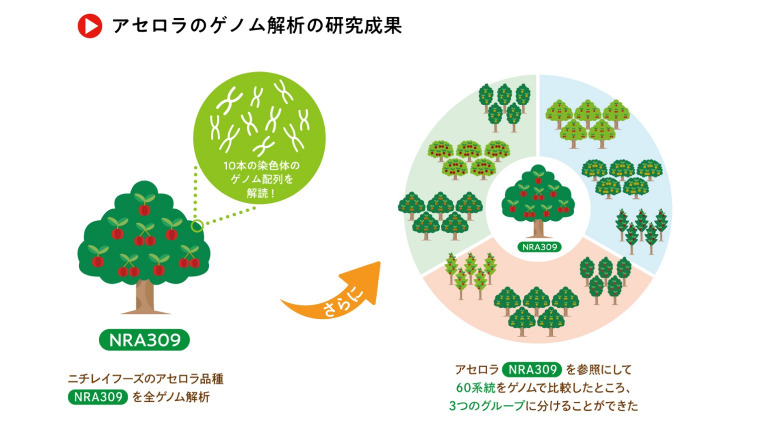
今回はニチレイフーズが開発した、果実の天然ビタミンC含量が高い「NRA309」というアセロラの品種の全ゲノム解読が行なわれた。
このゲノム解析には、最新のDNA分析技術を利用したため、これまでほとんどゲノム情報の蓄積がなかったアセロラにおいても、10本の染色体に対応する塩基配列をほぼ完全に読み取ることができたという。
また、ゲノム解析の結果から、アセロラの60系統は3つのグループに分けられることを明らかにした。なお、これらの解析に用いた材料はニチレイフーズと鹿児島大学の共同研究により作出された。
研修結果の発表に際して、ニチレイフーズでは以下のようにコメントしている。
「今回のゲノム解読により、『NRA309』の配列がアセロラのスタンダードとなり、今後の研究開発が進みます。果実に含まれる天然ビタミンCやポリフェノールの量を増やすための品種改良や、安定した生育、高い機能性を保持したアセロラの開発などが期待されます。アセロラが含まれるヒイラギトラノオ属やキントラノオ科におけるゲノム研究にも貢献できることが考えられます」
タイトル:Chromosome-scale genome assembly of acerola (Malpighia emarginata DC.)
著者:Kenta Shirasawa, Kazuhiro Harada, Noriaki Haramoto, Hitoshi Aoki, Shota Kammera, Masashi Yamamoto, Yu Nishizawa
掲載誌:DNA Research
DOI: https://doi.org/10.1093/dnares/dsae029
本研究は、(公財)かずさDNA研究所、JSPS科研費(22H05172、22H05181)の研究助成を受けたものだ。
本研究のポイント
・世界初アセロラの全ゲノム解析をニチレイフーズ登録NRA309で解読
・アセロラのゲノム解析が60系統まで解読
・今後のアセロラの品種改良や機能性付与の研究が促進に期待
関連情報
https://www.nichireifoods.co.jp
構成/清水眞希















 DIME MAGAZINE
DIME MAGAZINE













